NGS(RNA-Seq)
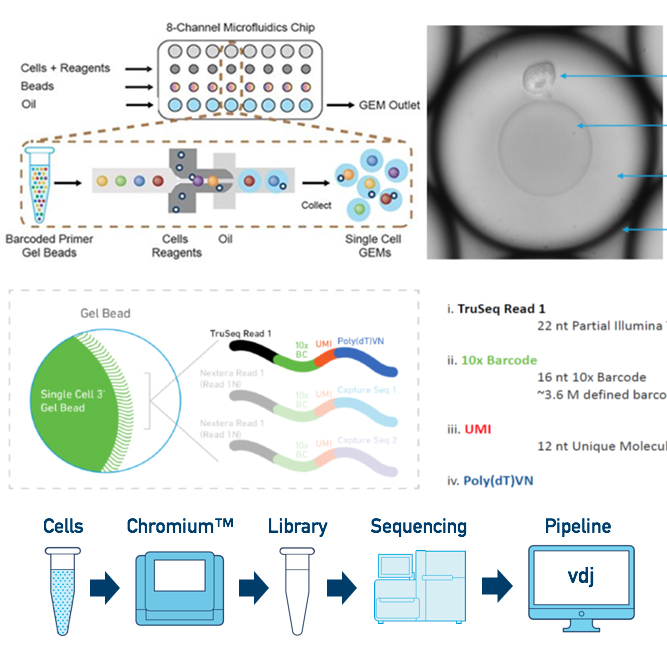
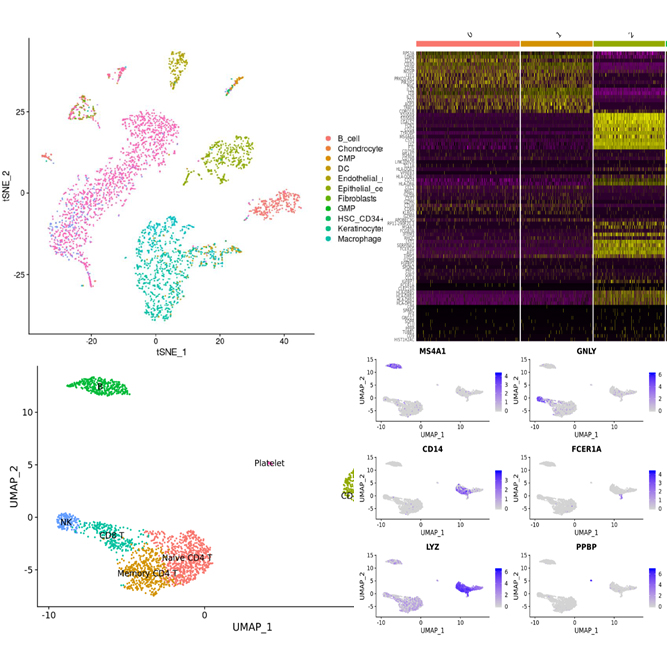
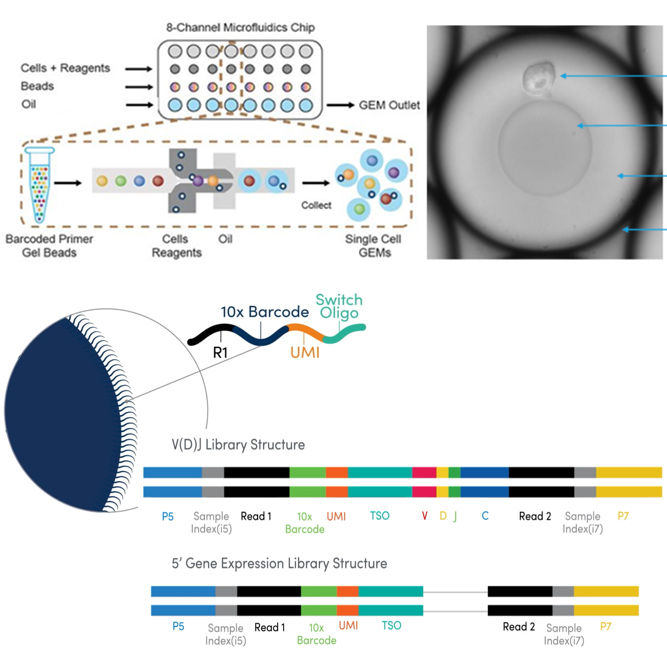
조직이나 세포 집단의 전사체 분석의 한계점을 극복하기 위해 단일세포의 전사체를 분석할 수 있는 Single cell RNA-Seq 서비스를 제공함으로써 세포간 유전자 발현의 다양성과 세포 타입별 identity를 확인할 수 있으며 UMAP, Feature selection, Clustering 등 다양한 분석을 지원해 드리고 있습니다.
Service Info.
| Sample requirement++ | - Cell line : ~1x106 cells in 14ml media(Cell viability >90%) - Primary cell or FACS sorted cell : 250,000 cells in 500 ul media(Viability >70%) |
|---|---|
| Library method | 10X Genomics Next GEM technology |
| NGS run format | Nova-Seq 6000, PE100bp |
| Data yield++ | ~60,000 reads(30,000 Paired Reads) per cell | Number of Target cell++ | ~5,000 cells |
| Turnaround time | ~4 weeks after Cell counting & QC |
| Sample type | Cell, Nuclei |
++ 초기 세포수, 목표 세포수, 시퀀싱 read수는 서비스 의뢰시 연구자와 상의하여 조정이 가능합니다.
- Cell Count & QC
- Library construction
- NGS
- Data Analysis
- Report
Data Analysis
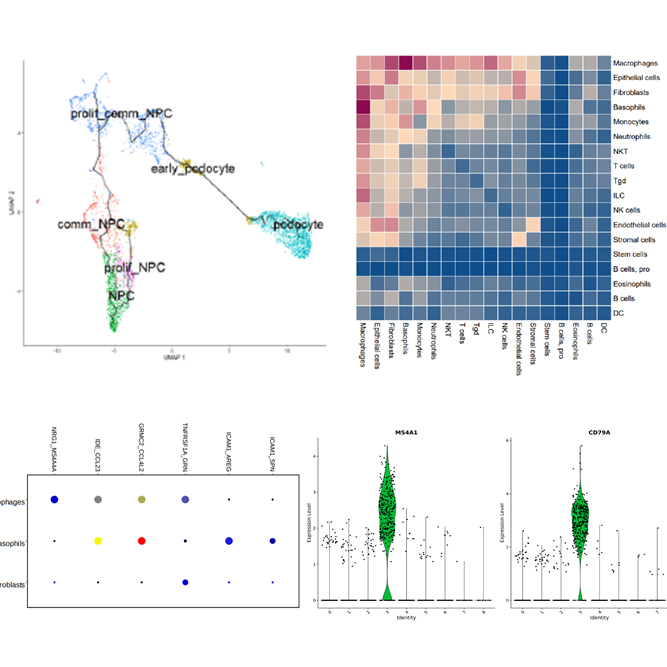
QC report와 Cell Ranger와 Loupe/Seurat 프로그램을 이용한 Gene expression data, UMAP, tSNE, Feature selection, Clustering heatmap, PCA, Cluster biomarkers, Cell type annotation, Trajectory inference, Cell-Cell interaction 등의 다양한 분석을 지원해 드립니다.
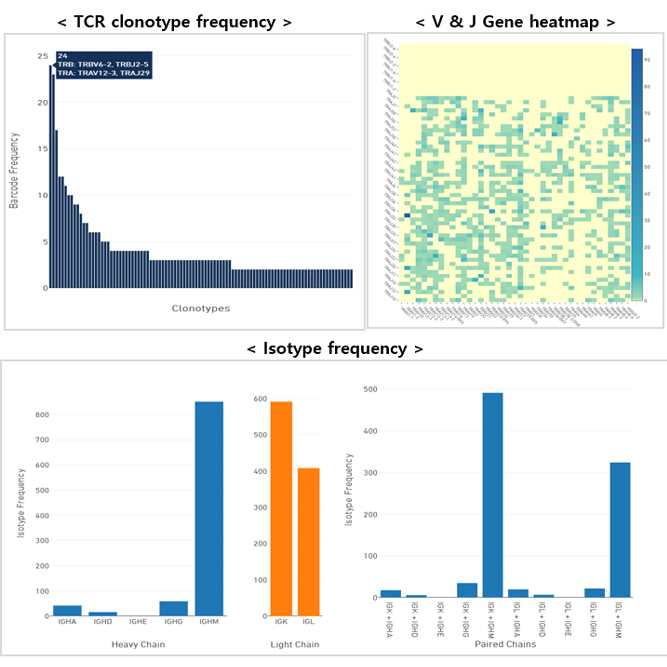
단일 세포의 해상도에서 전사체의 발현과 면역계의 세포 이질성, T 세포와 B 세포의 다양성 뿐만 아니라 항원 특이성을 동시에 확인하여 Immune system의 세포적 맥락을 파악할 수 있으며, UMAP, Feature selection, Violin Plot등의 분석과 더불어 Cell type별 clonotype 판별, TCR과 BCR에 대한 Clonotype frequency, Full-length V(D)J Gene sequences와 구성 유전자, Heavy-Light chain & α-β chain pairing 등 다양한 분석을 지원해 드리고 있습니다.
Service Info.
| Sample requirement++ | - Cell line : ~1x106 cells in 1.5ml tube with media (Cell viability >90%) - Primary cell of FACS sorted cell : 250,000 cells in media (Cell viability >70%) |
|---|---|
| Library method | 10X Genomics Next GEM technology |
| NGS run format | Nova-Seq 6000, PE100bp |
| Data yield++ | * Minimum 15,000 reads pairs per cell for V(D)J library * Minimum 50,000 reads pairs per cell for 5’ Gene Expression library |
Number of Target cell++ | ~10,000 cells |
| Turnaround time | ~4 weeks after Cell counting & QC |
| Sample type | Cell |
++ 초기 세포수, 목표 세포수, 시쿼싱 read수는 서비스 의뢰시 연구자와 상의하여 조정이 가능합니다.
- Cell Count & QC
- Library construction
- NGS
- Data Analysis
- Report
Data Analysis
QC report와 Cell Ranger와 Loupe/Seurat 프로그램을 이용한 Gene expression data, UMAP, t-SNE, Feature selection, Clustering heatmap, PCA, Cell type annotation, Cell type별 clonotype 판별, TCR과 BCR의 Clonotype frequency, Full-length V(D)J Gene sequences와 구성 유전자, Heavy-Light chain & α-β chain pairing 등의 다양한 분석을 지원해 드립니다.
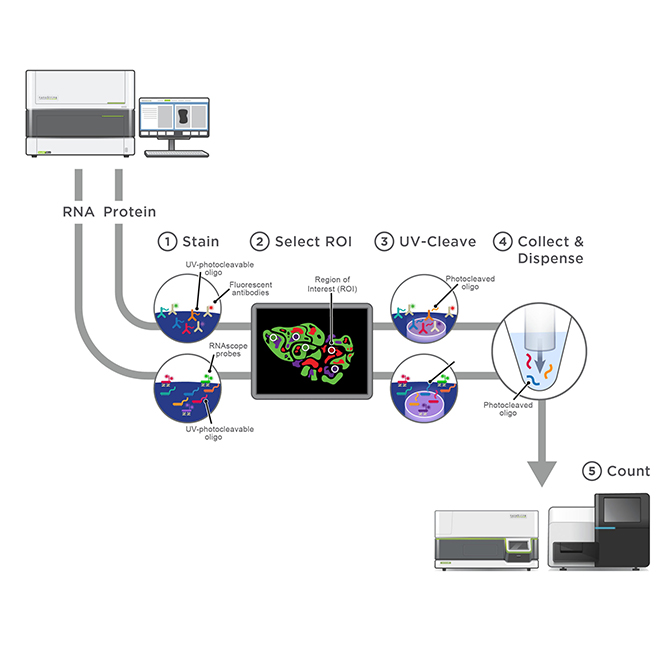
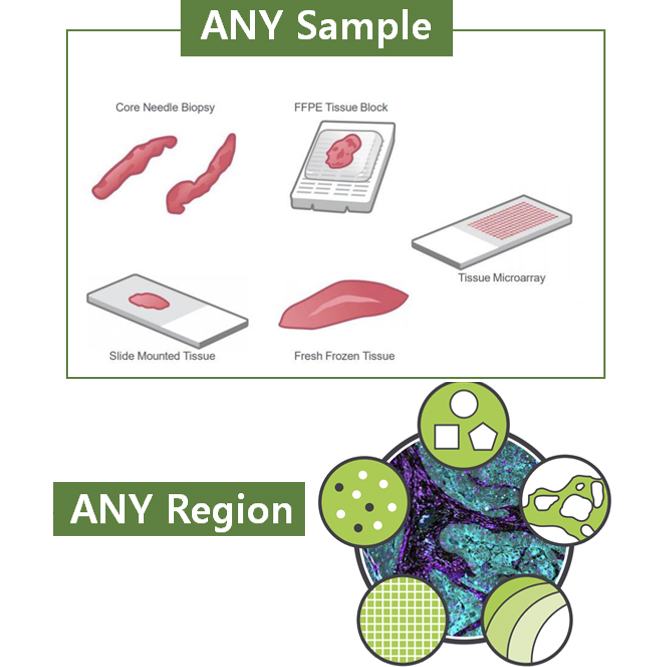
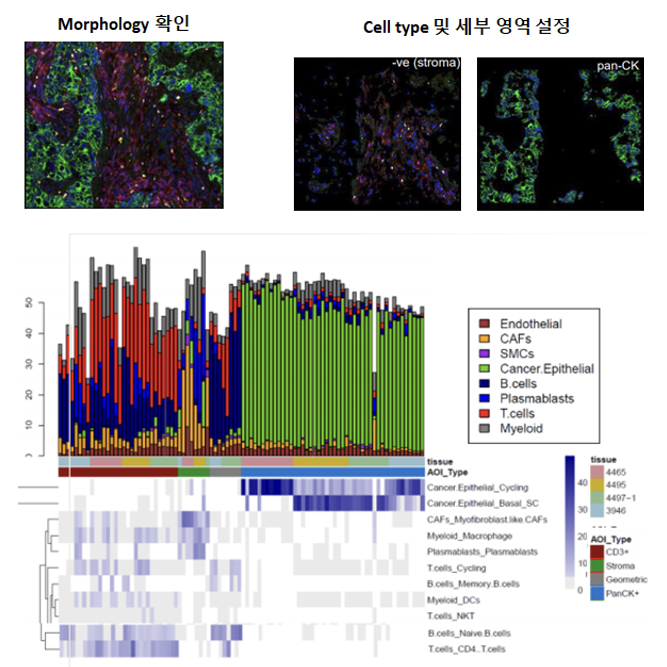
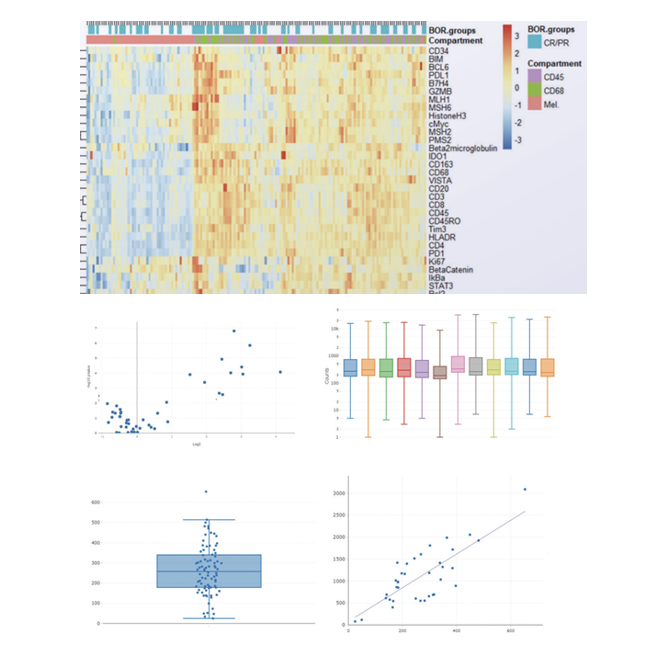
GeoMx DSP장비를 이용하여 조직 내에서 면역염색을 통해서 세포의 morphology 를 확인하고 원하는 영역, 세포 타입에 대한 유전자 발현값을 확인할 수 있는 서비스 입니다. Fresh Frozen, FFPE sample type에 상관없이 Human / Mouse의 모든 조직에서 실험 가능한 장점을 가지고 있으며, Deconvolution, PCA, tSNE, UMAP 등 다양한 분석을 지원해드리고 있습니다.
Service Info.
| Service Name | GeoMx Spatial WTA* Service | GeoMx Spatial CTA* Service |
|---|---|---|
| Species | Human, Mouse | Human |
| Number of Gene | Human ~18,000개 / Mouse ~20,000개 | ~1,800개 |
| Data yield | ~100 reads/㎛2 | ~30 reads/㎛2 |
| Sample type | Tissue (FFPE, Tissue microarray, Fresh frozen tissue, Core needle biopsy etc,.) | Sample size | 14.6 x 36.2 mm |
| Turnaround time | ~3 weeks after ROI selection | |
| Publication | NanoString GeoMx Publication | |
*WTA (Whole Transcriptome Atlas) / *CTA (Cancer Transcriptome Atlas)
- Service Consulting
- Staining
- ROI Selection
- Library Construction & NGS
- Report
Data Analysis
Gene expression 정보와 spatial information을 결합한 분석을 지원해드립니다. 또한 clusters, heatmaps, volcano plots, bar graphs, box plots, scatter plots과 같은 다양한 visualization이 가능합니다.
Genome Sequencing이 완료된 생물종을 대상으로 RNA를 분리하여 NGS 기술을 통해 전사체(transcriptome) 분석을 수행해 드립니다. mRNA expression profiling 분석 이외에도 lncRNA, isoform, INDEL, Alternative Splicing Event, Gene fusion 등 다양한 분석을 지원합니다.
Service Info.
| total RNA-Seq | mRNA-Seq | |
|---|---|---|
| Sample requirement | >2ug total RNA | >2ug total RNA |
| Library method | RiboCop rRNA Depletion kit + CORALL RNA-seq Library Prep kit |
Poly(A) RNA Selection kit + CORALL RNA-seq Library Prep kit |
| NGS run format | HiSeq or NovaSeq, PE100 | HiSeq or NovaSeq, PE100 |
| Data yield | >6Gb/sample | >4Gb/sample |
| Turnaround time | ~4 weeks after RNA QC | ~4 weeks after RNA QC |
| Sample type | Tissue, Cell, Whole Blood(Paxgene), Plant, Fungi, Bacteria, etc. | |
++ RNA prep.을 원하실 경우 담당자와 상의하여 진행이 가능합니다.
- RNA QC
- Library prep.
- NGS
- Data Analysis
- Report
Data Analysis
| Basic Analysis | Optional Analysis | |||||
|---|---|---|---|---|---|---|
| Lnc RNA | mRNA Expression | Isoform Expression | INDEL/SNP Analysis | Splicing Variant | Gene Fusion | |
| Total RNA-Seq |
O | O | O | O | O | O |
| mRNA-Seq | O | O | O | O | O | |
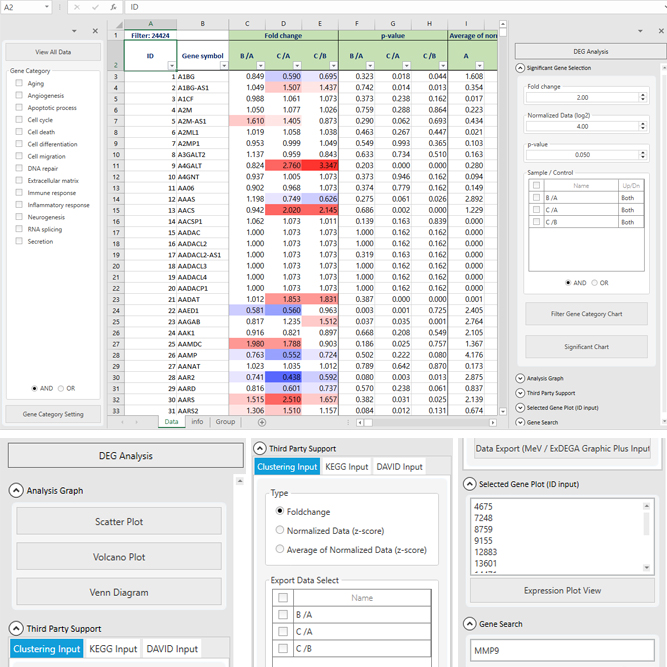
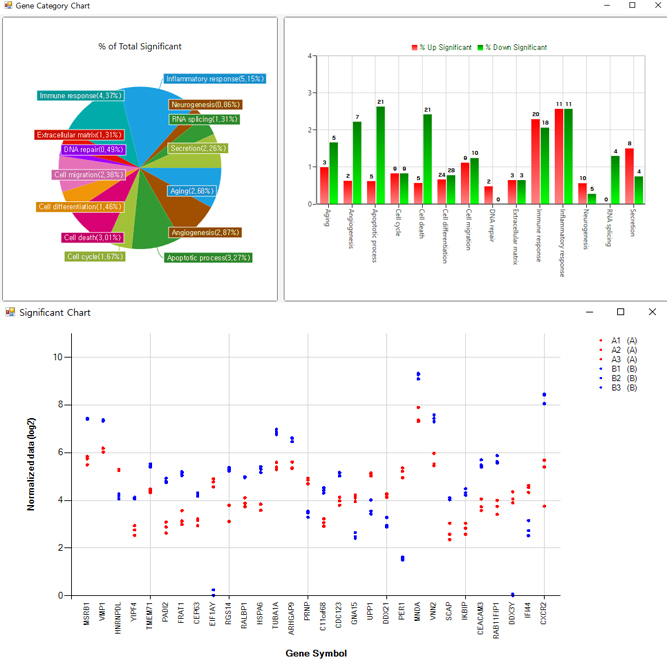
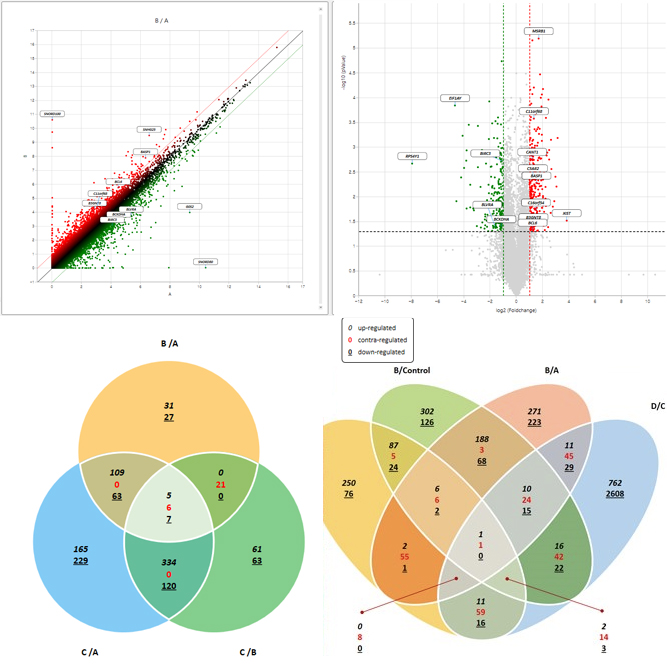
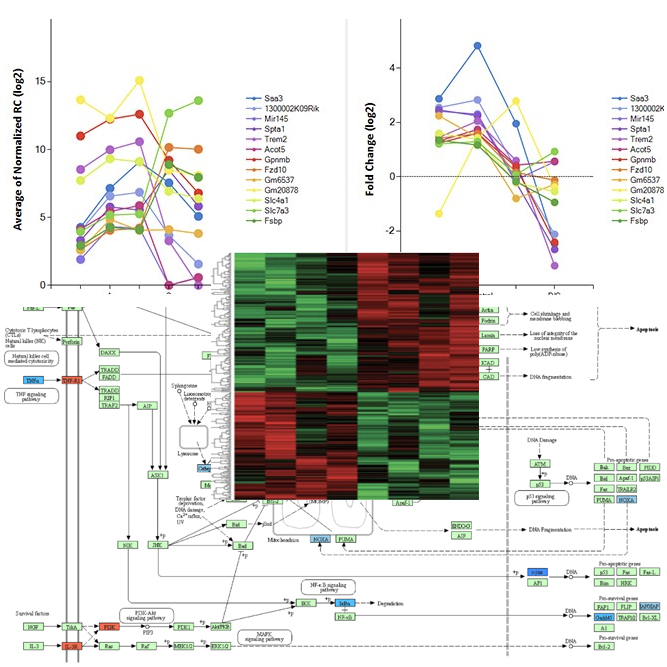
ExDEGA Report, GO, Pathway, Clustering, Network, GSEA, Data Mining 등의 다양한 분석을 지원해 드립니다.
Poly(A)가 존재하는 생물종을 대상으로 RNA를 분리하여 3’ UTR 부분을 짧게 시퀀싱을 수행하고 유전자 발현 분석을 수행해 드립니다. mRNA expression profiling 분석만을 요구할 시 매우 저렴하게 고품질의 데이터를 제공해 드립니다.
Service Info.
| Sample requirement | >10ng total RNA |
|---|---|
| Library method | QuantSeq 3' mRNA-Seq Library Prep Kit FWD |
| NGS run format | NextSeq 500/550, SE 75bp |
| Data yield | >10M read/sample |
| Turnaround time | ~3 weeks after RNA QC |
| Sample type | Tissue, Cell, Whole Blood(Paxgene), Plant, Fungi, FFPE, Sorted cell, etc. |
++ RNA prep.을 원하실 경우 담당자와 상의하여 진행이 가능합니다.
- RNA QC
- Library prep.
- NGS
- Data Analysis
- Report
Data Analysis
ExDEGA Report(mRNA expression), GO, Pathway, Clustering, Network, GSEA, Data Mining 등의 다양한 분석을 지원해 드립니다.
Bacteria RNA-Seq은 total RNA에서 rRNA만을 효율적으로 제거하는 것이 Data quality에 중요한 요소로 작용하고 있습니다. 이바이오젠에서는 박테리아의 rRNA에 single-stranded DNA probes를 binding시켜 효과적으로 hybridized rRNA를 제거한 후, 박테리아 mRNA만을 분리하여 NGS 기술을 통해 전사체(transcriptome) 분석을 수행해 드립니다.
Service Info.
| Sample requirement | >2ug total RNA |
|---|---|
| Library method | RiboCop rRNA Depletion kit + CORALL RNA-seq Library Prep kit |
| NGS run format | HiSeq or NovaSeq, PE100(Paired End 100bp) |
| Data yield | >~2Gb (Data yield is varied depending on the species) |
| Turnaround time | ~4 weeks after RNA QC |
| Available species | NCBI DB에 등록되어 genome sequencing이 완료된 prokaryotes에 해당하는 박테리아종 (Species Search) |
++ RNA prep.을 원하실 경우 담당자와 상의하여 진행이 가능합니다.
- RNA QC
- Library prep.
- NGS
- Data Analysis
- Report
Data Analysis
ExDEGA Report(mRNA expression), GO, Pathway, Clustering, Network, GSEA, Data Mining 등의 다양한 분석을 지원해 드립니다.
세포, 조직, exosome, body fluid 등에 존재하는 microRNA, piRNA와 같은 small size의 RNA의 양을 NGS 시퀀싱을 통해 분석하고 해당 데이터베이스 기반으로 target gene 분석 및 다양한 데이터 분석을 수행합니다.
Service Info.
| Sample requirement | >2ug total RNA (Tissue, Cell) >20ng RNA (Exosome, Bodyfluid) |
|---|---|
| Library method | NEBNext Multiplex Small RNA Library Prep Kit |
| NGS run format | NextSeq 500/550, SE 75bp |
| Data yield | >20M read/sample |
| Turnaround time | ~4 weeks after RNA QC |
| Sample type | Tissue, Cell, Whole Blood(Paxgene), Serum, Plasma, Urine, Exosome, etc. |
++ RNA prep.을 원하실 경우 담당자와 상의하여 진행이 가능합니다.
- RNA QC
- Library prep.
- NGS
- Data Analysis
- Report
Data Analysis
ExDEGA Report(miRNA expression), piRNA profiling, GO, Pathway, Clustering, Network, GSEA, Data Mining 등의 다양한 분석을 지원해 드립니다.